タンパク質の設計図を壊さないようにDNA切断を修復する新たな仕組みを発見 ~がんの発生を巧妙に抑制するメカニズムの一端を解明~
群馬大学(群馬県前橋市)未来先端研究機構 柴田淳史准教授らは、東京大学大学院医学系研究科、米国マサチューセッツ総合病院等とともに、特定のタンパク質が、DNA修復中に形成された不安定なR-loop構造の崩壊を防ぎ、DNA切断を正確に修復する新たな仕組みを発見しました。これにより、タンパク質の設計図の異常を原因として生じるがんなどの疾患を防ぐために細胞が持っている防御メカニズムの一つを解明しました。
本件のポイント
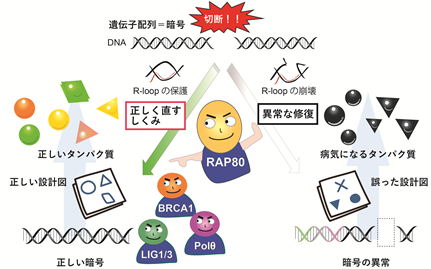
- タンパク質の設計図が記録されたDNAが切断(注1)された場合に、読み出されていたRNA(注2)が、DNA切断部位と近い場所でDNAと結合してR-loopと呼ばれる不安定な構造(注3、図1)ができてしまい、それが崩壊すると、設計図が壊れてしまうことがわかった。
- DNA修復タンパク質であるRAP80(注4)はこの不安定な構造を崩壊から守り、正確なDNA切断の修復を誘導することがわかった。
- RAP80の量が少ない細胞では、がんの発生につながるようなタンパク質設計図の異常が高頻度で見つかったことから、今回発見したメカニズムは、がんの発生を抑制するために細胞が持つ巧妙な防御機構の一つであることが示唆された。
本件の概要
私たちの体のほとんどの細胞は、タンパク質の設計図としてコンパクトに折りたたまれたDNAを持っています。一つの細胞がもつDNAの長さは2メートルほどになると言われていますが、そのうちのほんの一部分しか設計図として利用されていません。設計図がRNAとして読み出されている部分は、細胞にとって非常に重要であるため、DNA切断などが起こった場合でも正確に修復して維持することが重要です。実際、設計図が壊れて変化してしまうと、大切なタンパク質が作られなくなったり、異常なタンパク質が作られたりすることで、がんなどの病気が発生してしまいます。設計図が頻繁に読み出されている部分、つまりRNAがたくさん存在する部分でDNA切断が起こると、RNAとDNAが対合することで、R-loopと呼ばれる構造が形成されることが知られていました。普段は、DNAは二本鎖で非常に安定に存在しますが、R-loop構造のように、DNAが一本鎖になる部分があると、DNAを切断するタンパク質がアクセスしやすくなってしまい、DNAを壊しやすくなってしまいます。従って、R-loop構造は非常に不安定な構造といえます。しかしながら、DNA切断を修復しなければならない状況にも関わらず、細胞がなぜこの不安定なR-loop構造の形成を許容し、またどのように巧妙に処理して正確なDNA切断の修復につなげているかはわかっていませんでした。
今回、東京大学大学院医学系研究科の安原崇哲助教、加藤玲於奈大学院生、宮川清教授、マサチューセッツ総合病院のLee Zou教授、群馬大学の柴田淳史准教授らの国際共同研究グループは、タンパク質RAP80が、DNA修復中に形成された不安定なR-loop構造の崩壊を防ぐこと、さらにRAP80が機能しないと、不安定なR-loop構造の崩壊に伴ってタンパク質の設計図に異常が増えてしまうことを発見しました。RAP80によって保護されたR-loop構造は、BRCA1、Polθ、 LIG1/3を介してDNA切断を正確に修復することも明らかとなりました。今回明らかになったメカニズムは、タンパク質の設計図の異常を原因として生じるがんなどの疾患を防ぐために細胞が持っている防御機構の一端であることが示唆されます。
本研究成果は、米国科学雑誌『Cell Reports』の2022年2月1日 オンライン版に掲載されます。
研究内容
DNA損傷は日々我々のゲノムを脅かしており、損傷の中でも特にDNA二重鎖切断(DSB)は重篤なゲノム異常を誘導するため、正確に修復する必要があります。ヒト細胞においてDSBは、主に非相同末端結合と相同組換え修復機構の2つで修復されることが知られていますが、それらの間の経路選択は長年の議論の的となっていました。近年の研究で、DSB周辺での転写の活性化度合や、RNAそのものがDSBの相同組換え修復を推進することなどが明らかとなっています。また、DSB周辺に存在するRNAは、DNAとハイブリッド二重鎖構造を形成してR-loopを構築することで、DSB修復に影響を与えることが知られています。
一般に、一重鎖DNA領域はヌクレアーゼによる切断の標的となりやすいことから、一重鎖DNA領域を含むR-loop構造はゲノム不安定性の原因となります。DSB修復の過程ではヌクレアーゼがDSB末端を整えることで末端の結合をスムーズに行わせることが知られているため、R-loop構造がそれらのDNA分解酵素から保護されているか、もし保護されているとすればどのような仕組みで保護されているのかについて着目して研究を進めました。
今回、東京大学大学院医学系研究科の安原崇哲助教、加藤玲於奈大学院生、宮川清教授、マサチューセッツ総合病院のLee Zou教授、群馬大学未来先端研究機構の柴田淳史准教授らの国際共同研究グループは、転写共役型DSB修復メカニズムに関わる新たな因子RAP80を同定し、RAP80がDSB後に形成されるR-loop構造を分解から保護していることが明らかとなりました。RAP80が保護したR-loop構造は、その後BRCA1、Polθ、LIG1/3を介した正確なDSB修復機構である転写共役型末端結合を促進しており、これらの機構によって転写活性化領域のDSBを正確に修復していることがわかりました。RAP80が機能しない場合には、CtIPと呼ばれるDNA分解促進因子がDSB部位に過剰に蓄積し、R-loop構造の一重鎖DNA部位を分解してしまうことが判明しました。このようなR-loop構造の分解は、遺伝情報の欠失や、遺伝子融合などのゲノム異常を増加させることも明らかとなりました。従って、今回明らかになったRAP80を介するR-loop構造の安定性の維持機構は、我々のゲノムの最も重要な部位の遺伝子情報を正確に維持し、がんなどで頻繁に見られるようなゲノム異常の発生を抑制するために細胞が持つ根源的なメカニズムの一つであると考えられます。
発表雑誌
- 雑誌名:Cell Reports 2022年2月1日 オンライン版
- 論文タイトル:RAP80 suppresses the vulnerability of R-loops during DNA double-strand break repair
- 著者:Takaaki Yasuhara*†, Reona Kato†, Motohiro Yamauchi, Yuki Uchihara, Lee Zou, Kiyoshi Miyagawa*, Atsushi Shibata** (†co-first author, *corresponding authors, **lead contact)
- DOI番号: 10.1016/j.celrep.2022.110335
本件に関するお問合せ先
研究に関すること
群馬大学 未来先端研究機構 DNA修復学研究室
准教授 柴田 淳史 (しばた あつし)
Tel: 027-220-7977 Fax:027-220-7909
E-mai:shibata.at@gunma-u.ac.jp
柴田研究室ホームページ
東京大学大学院医学系研究科 附属疾患生命工学センター 放射線分子医学部門
助教 安原 崇哲 (やすはら たかあき)
教授 宮川 清 (みやがわ きよし)
Tel:03-5841-3505 Fax:03-5841-3013
E-mail:tyasuhara-tky@umin.ac.jp; miyag-tky@umin.ac.jp
報道に関すること
群馬大学総務課広報係
TEL:027-220-7010 FAX:027-220-7012
E-mail:s-public@ml.gunma-u.ac.jp (係共通)
東京大学医学部・医学系研究科総務チーム
TEL:03-5841-3304 FAX:03-5841-8585
E-mail:ishomu@m.u-tokyo.ac.jp (係共通)
用語解説
(注1)タンパク質の設計図が記録されたDNAが切断
我々の遺伝情報、つまりタンパク質の設計図は暗号化されてDNAとして記録されており、体内のほぼ全ての細胞が個々に保持している。DNAはさまざまな外的、内的ストレスによって日々損傷を受けていることが知られている。外的ストレスの代表的なものは、放射線、紫外線、化学物質などがあり、内的ストレスの代表的なものは、細胞内の代謝の過程で発生する複製のミスや、酸化ストレスなどがある。細胞内にはこれらの損傷に対応するための仕組みが多数備わっており、遺伝情報に異常が生じないようにしている。本研究では、ゲノム損傷の中でも、DNA二重鎖が完全に切断されてしまうという最も重篤な損傷に対応するメカニズムを解明した。
(注2)RNA
DNAに暗号として記録されたタンパク質の設計図は、まずRNAとして読み出される。この読み出しの過程のことを「転写」と呼ぶ。その読み出された設計図、つまりRNAの情報を基にして、タンパク質へと翻訳していく。タンパク質とは様々な種類のアミノ酸が一列に並んだものの総称であり、それぞれのタンパク質に固有のアミノ酸の配列が、RNAに記録されている。
(注3)R-loopと呼ばれる不安定な構造
RNAはDNAを基にして読み出されるため、容易に対合してRNAとDNAからなる二重鎖になる。これがDNA二重鎖の中の一部に形成される、つまり、元々DNA-DNAで対合していた二重鎖がRNA-DNA二重鎖に置き換えられた場合には、2本のDNAのうちの1本が対合する相手を失い、一重鎖DNAとして存在するようになる。このような三重鎖からなる構造は、R-loop(Rループ)と呼ばれ(図1)、我々のDNA二重鎖の中にもこのような構造を作りやすい部分が多数存在することが知られている。一重鎖DNAは、DNAを切断するタンパク質(ヌクレアーゼ)によって容易に切断されてしまうため、一般にR-loopは不安定な構造であると考えられている。RNAが豊富に存在する部位でDNA切断が起こった場合には、周辺でR-loop構造が形成されてしまうため、それらの構造が切断されてしまうのを防ぐための仕組みが必要となる。
(注4)RAP80
DNA修復タンパク質であるRAP80はDNAの切断の発生を細胞全体に知らせるシグナルを媒介する因子として知られていた。しかしながら、今回の研究ではRAP80がDNA切断の修復中に、不安定なR-loop構造を保護し、R-loop構造内に含まれる一重鎖DNAが切断されてしまうのを防ぐという、全く予想外の機能が明らかとなった。
添付資料
本来はDNA二重鎖で我々の遺伝情報が保持されているが、ある特別な環境下では、DNA-RNAハイブリッドによる二重鎖と、一重鎖DNAによって構成されるR-loop構造が形成され、その構造の存在がさまざまな細胞機能に影響することが近年明らかになりつつある。
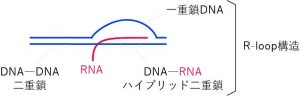
図1 R-loop構造の模式図
プレスリリース
タンパク質の設計図を壊さないようにDNA切断を修復する新たな仕組みを発見 ~がんの発生を巧妙に抑制するメカニズムの一端を解明~
